R言語 plotの続き。
ビジュアル的にはggplot2パッケージを使ったほうが良いが、plotと少し違うので最初は戸惑う。
基本的な作業の流れは、
library(ggplot2)でggplot2パッケージを有効化して
用意したdataを
ggplot(data, aes関数) + geom_○○関数で描画する。
※ ggplot2パッケージのインストールは、
install.packages("ggplot2") と入力するか
パッケージ - パッケージのインストール
で表示されるウィンドウから選択する
描画
・ まず用意したデータファイル .csvをread.csv()で読み込む。
現在進行形の新型コロナウイルス感染者数の推移 covid19.csv。
data <- read.csv("covid19.csv",header=T)1列目(DAY)の日付をDate型に変換。
data[,1] <- as.Date(data[,1],"%Y/%m/%d")ここまでは前回のplotの時と同じ。
・ ggplot2で描画。
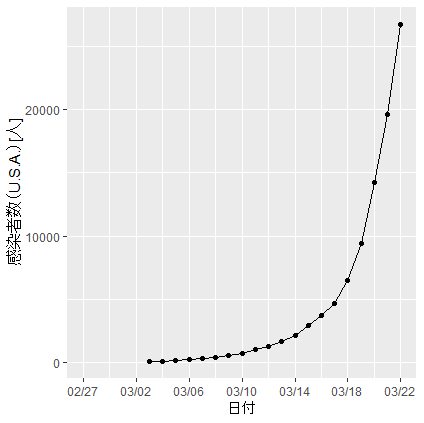
library(ggplot2)U.S.A. のデータを描画してみる。
ggplot(data,aes(data[,1],data[,9])) + geom_point()で点々(離散プロット)。
ggplot(data,aes(data[,1],data[,9])) + geom_line()で折れ線グラフ。
折れ線グラフ + 点々で描画してみる。pのところは任意。
p <- ggplot(data,aes(data[,1],data[,9])) + geom_line() + geom_point()+ X軸、Y軸のラベル付け替え
p <- p + xlab("日付") + ylab("感染者数(U.S.A.)[人]")もしくは
p <- p + labs(x="日付", y="感染者数(U.S.A.)[人]")でも同じ。
前回のplotの際xaxtやaxis.Date()で対処したX軸の日付表示は、scale_x_date()を使うようだ。at=…がbreaks=…、format=…がdate_labels=…に対応している。
p <- p + scale_x_date(breaks = function(x) seq.Date(from=data[1,1],to=max(x),by="4 days"),date_labels="%m/%d")で4日ごとの目盛。
最後に
pで描画。
イマージェンシー
追)軸の目盛の調整例
X軸 2000年-2020年 2年ごとの目盛
p <- p + scale_x_continuous(breaks = seq(2000,2020,by=2))多重描画
複数の国々のデータを重ね合わせて描画してみる。
データの加工が必要だが、一括で描画してくれる。
Excel等で加工する。
※ dplyrパッケージもある
感染者数を1列(NUMBER)に並べる。
以下加工後のイメージ。
※ 一応covid19-2.csvファイルは、こちらに保存。詳細データは大元へ
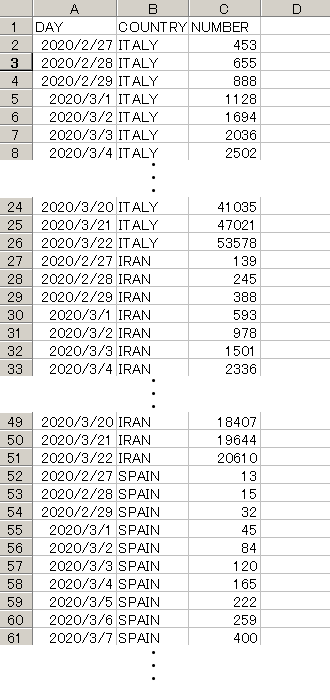
data2 <- read.csv("covid19-2.csv",header=T)
data2[,1] <- as.Date(data2[,1],"%Y/%m/%d") 折れ線グラフ + 点々で描画。qのところは任意。
colour=COUNTRYで色分け。
q <- ggplot(data2,aes(DAY,NUMBER,colour=COUNTRY))
q <- q + geom_line(size=1) + geom_point()
q <- q + labs(x="日付", y="感染者数[人]", colour="国")
q <- q + scale_x_date(breaks = function(x) seq.Date(from=data2[1,1],to=max(x),by="4 days"),date_labels="%m月%d日")
q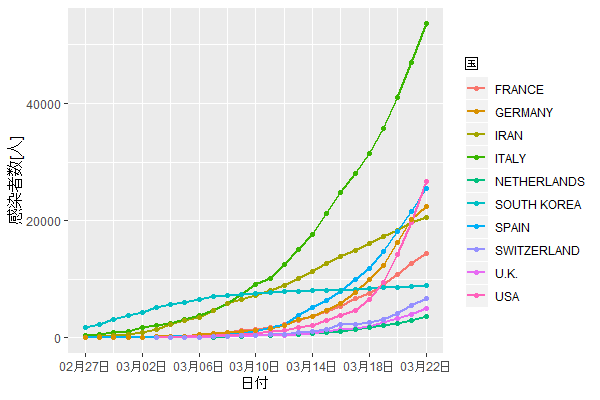
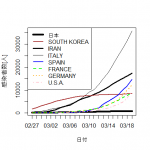
日本は1000人超。
10グループもあると識別しにくい。7グループ以上で青緑系が識別しにくくなる。
残念ながらイタリアが未だ鈍化の兆候がみられず酷い状況。
「医療関係者が感染 + 医療機関へ人が殺到」が、最悪パターン。
※ クルーズ船もおそらく乗員が感染して感染者が急増
イラン、フランスも深刻だが、スペイン、ドイツ、アメリカの増え方が急激。
一難去った後も嵐の予感。